药学院侯廷军团队在Research报道基于AI的共价可靶半胱氨酸预测方法
共价抑制剂(TCI)与靶标的结合亲和力强,在结合位点具有较长的保留时间,有望解决部分靶标的选择性与不可靶向性的问题,也因此共价抑制剂受到了越来越多的关注。共价抑制剂与靶标的相互作用既包含非共价部分的非键相互作用,也包括共价弹头与结合口袋亲核氨基酸之间的共价成键作用。然而,对共价结合位点的认识非常有限,对于共价抑制剂的研究也局限在很少部分的靶标中。因此,开发准确、快速分析蛋白质结构中氨基酸共价可靶性的方法,可以扩大共价抑制剂的研究范围,加速共价药物的发现。

2022年7月,浙江大学药学院侯廷军教授/潘培辰研究员团队在Research杂志在线发表了题为“Proteome-Wide Profiling of the Covalent-Druggable Cysteines with a Structure-Based Deep Graph Learning Network”的研究论文,提出新型基于图表示的深度学习方法DeepCoSI用于共价药物结合位点的预测。
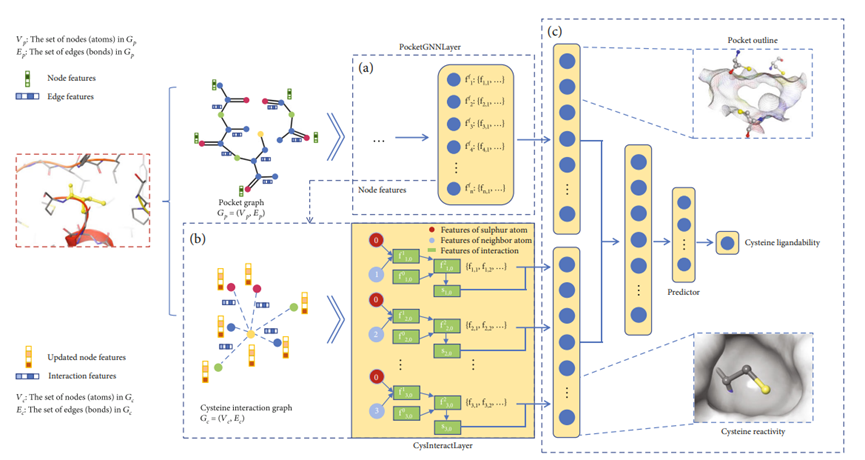
图 1 DeepCoSI模型结构框架
该模型由两个模块组成,通过结合物理化学特征和几何特征,分别对结合“口袋”结构性质和半胱氨酸的“内在反应性”进行表征,进而对半胱氨酸的共价可靶性进行预测。在两个外部测试集上的结果显示,模型具有很强的区分共价可靶半胱氨酸与其他半胱氨酸的能力。以归一化后的排序值(阳性样本排序/半胱氨酸数目)作为标准,当阈值设为0.5时,模型在两个测试集中的预测成功率分别可以达到98 %和82.9 %。最后,作者应用该模型将RCSB PDB中所有人源蛋白结构中的半胱氨酸进行了共价可靶性分析,研究人员可在共价抑制剂数据库CovalentInDB(http://cadd.zju.edu.cn/cidb/deepcosi/cys)中检索并使用这部分预测数据。
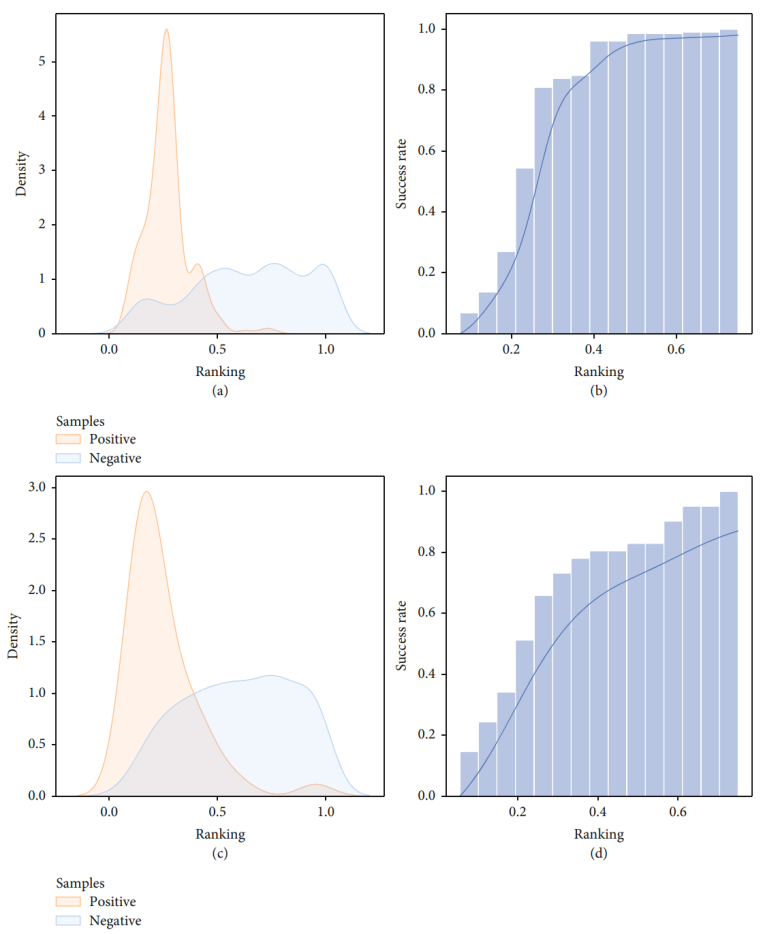
图 2 DeepCoSI在外部测试集上的表现
浙江大学药学院为本论文的第一署名单位,浙江大学药学院博士生杜红岩和蒋德军博士为共同第一作者,浙江大学药学院潘培辰研究员和侯廷军教授、中南大学曹东升教授为共同通讯作者。
原文链接:
https://spj.sciencemag.org/journals/research/2022/9873564/


