药学院范骁辉课题组在Nature Communications报道首个可将传统转录组测序数据重构至单细胞空间分辨率的方法
近年来,单细胞测序(scRNA-seq)和空间转录组测序(Spatially resolved transcriptomics)技术彻底变革了人们理解复杂组织的方式,不断推动生命健康研究进入单细胞和空间组学时代。然而,目前的单细胞测序及空间转录组技术不仅耗时费力,而且成本极为昂贵,严重限制了其推广应用。与此同时,传统混样测序(Bulk RNA-seq)一直是生物医药研究的主流工具,特别是TCGA、ENCODE、ICGC等多个多大型项目的相继开展为相关研究累积了海量数据资源。因此,如何充分利用原有数据资源,从Bulk数据中得到单细胞分辨的空间基因表达谱,是系统生物学研究的重大技术挑战。
2022年10月30日,浙江大学药学院、长三角智慧绿洲创新中心范骁辉教授团队联合计算机科学与技术学院陈华钧教授团队和军事医学科学院高月教授团队在国际知名期刊《Nature Communications》上发表了题为“De novo analysis of bulk RNA-seq data at spatially resolved single-cell resolution”的研究论文。该研究提出了一种空间解卷积算法——Bulk2Space,利用β-VAE等深度学习框架,首次实现将Bulk转录组重构至单细胞空间分辨率。
研究团队利用Bulk2Space揭示了不同肿瘤区域中B淋巴细胞的基因表达差异以及组织在炎癌转化过程中的空间转录异质性。本研究还将Bulk2Space应用于小鼠脑组织不同脑区Bulk数据的空间解卷积分析。相关数据均采自范骁辉教授团队自主研发的基于激光捕获显微切割技术(LCM)的高通量多样本空间测序技术——Spatial-seq。研究表明,Bulk2Space不仅可以重构小鼠皮层的层级空间结构,还可以对下丘脑中未知细胞类型进行二次注释。系列研究证实,Bulk2Space不仅在模拟和真实数据集中展现出了优异的性能,而且成功应用于多种生物学和疾病场景,具有广泛的应用前景。该算法现已开源至GitHub平台(https://github.com/ZJUFanLab/bulk2space)。
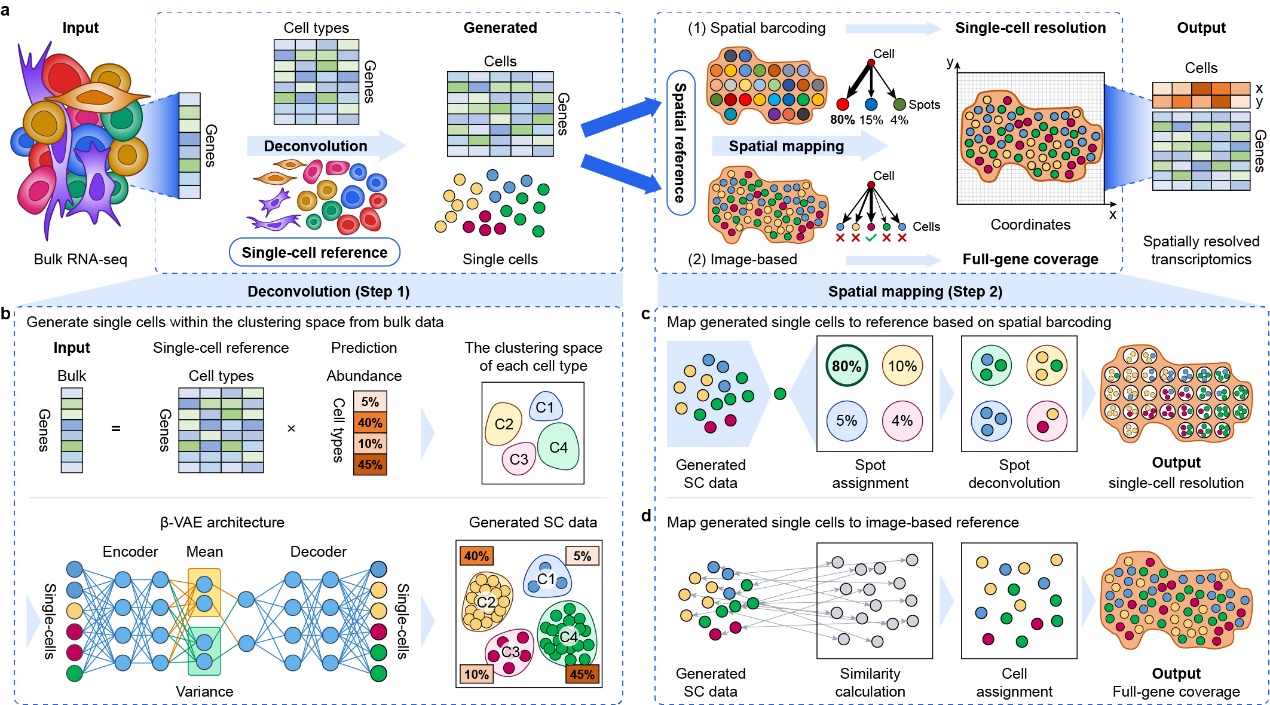
Bulk2Space算法原理示意图

利用Bulk2Space对Spatial-seq技术获得的小鼠下丘脑bulk转录组进行空间解卷积及细胞类型注释
本文第一作者为浙江大学药学院博士后廖杰、博士研究生钱竞扬以及浙江大学计算机科学与技术学院博士研究生方尹、陈卓和庄祥。通讯作者为浙江大学药学院范骁辉教授、浙江大学计算机科学与技术学院陈华钧教授和军事医学科学院放射与辐射医学研究所高月教授。本研究受到国家自然科学基金、国家中医药多学科交叉创新团队等项目资助以及阿里云支持。
原文链接:https://www.nature.com/articles/s41467-022-34271-z


